产品货号:
JN3052
中文名称:
Poly(A)聚合酶
英文名称:
E. coli Poly(A)Polymerase
产品规格:
100U
发货周期:
1~3天
产品价格:
询价
Poly(A)聚合酶是从重组菌株E.coli中分离得到,可以催化ATP以AMP的形式依次掺入到RNA的3'末端,即在RNA的3'末端加多聚A尾。Poly(A)聚合酶具有很高的加尾效率,可以在RNA的3'末端加入20~200个A碱基。聚腺苷酸化提高了RNA在细胞中的稳定性,增强了转染或显微注射后RNA的表达效率。Poly(A)尾在某些应用中可作为第一链cDNA合成的引物结合位点,并可用于末端标记或定量miRNA。

- 用于RNA 3'末端标记;
- 为RNA添加Poly(A)尾,用于克隆或亲和纯化。比如将miRNA添加Poly(A),为cDNA合成提供oligo-dT引物结合位点;
- 提高RNA在真核细胞中的翻译效率。

在37℃条件下,10min内将1nmol AMP掺入到RNA所需的酶量定义为1个活性单位(U)。

| 组分 | 规格 |
| Poly(A) Polymerase(5U/μL) | 20μL |
| 10×Poly(A) Polymerase Buffer | 1mL |
| ATP (10mM) | 100μL |
保存:-20℃

- 该酶只能以RNA为底物。
- 该酶将AMP添加到RNA的3'末端具有较高的选择性,并不会在所有RNA分子中添加相同长度的Poly(A)。
- 该酶需要Mg2+等二价阳离子才具有活性。
- RNA加A尾长度受酶量、ATP和反应时间等因素影响,不同的实验需要加A的量会有所不同,可通过控制反应时间及酶量来调整加A的长度,该酶在37℃反应30分钟时可加约30个A碱基,1小时可加约100个A碱基。
- 相同RNA量及反应时间随酶量增加,加尾长度增大,如下图:
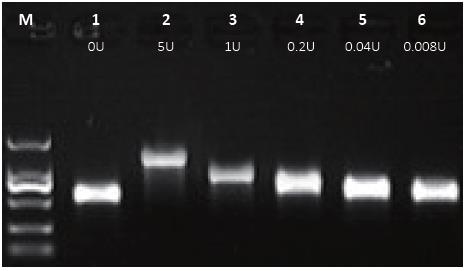
- 相同RNA量,加入5U的酶量,随时间延长,加尾长度逐渐增大,如下图:
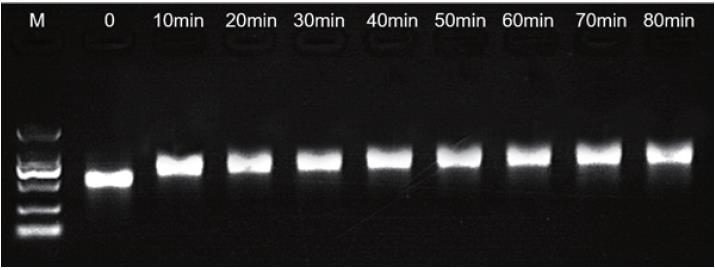
- 相同RNA量及反应时间随酶量增加,加尾长度增大,如下图:
- 该酶使用M-MuLV逆转录酶反应缓冲液,也可以进行反应。
- EDTA抑制该酶活性。若停止反应,可通过添加EDTA至终浓度10mM,或直接对反应体系进行纯化。
- 加尾结束后,在转染细胞或显微注射前,必须对RNA进行纯化,具体纯化方式见操作流程。

- 加尾流程
- 按下表加入各成分配制反应体系(20μL):
成分 用量 10×Poly(A) Polymerase Buffer 2μL ATP (10mM) 1μL RNA 1~10μg Poly(A) Polymerase(5U/μL) 0.2~1μL RNase-Free Water 至20μL - 充分混匀,37℃反应30~60min。
- 按下表加入各成分配制反应体系(20μL):
- RNA纯化
- 酚/氯仿纯化法
酚/氯仿抽提可去除蛋白和大部分游离核苷酸。- 加入160μL RNase-free Water将产物稀释至180μL。
- 加入20μL 3M的醋酸钠(pH5.2)到稀释后的产物中,用移液器充分混匀。
- 加入200μL的酚/氯仿混合液(1:1)进行抽提,室温10000rpm离心5min,将上层溶液(水相)转移至新的RNase-free EP管中。
- 加入与水相等体积的氯仿抽提2次,收集上层水相。
- 加入2倍体积的无水乙醇并混匀,-20℃孵育至少30min,4℃ 15000rpm离心15min。
- 弃上清并加入500μL预冷的70%乙醇洗涤RNA沉淀,4℃ 15000rpm离心,弃上清。
- 开盖干燥2min,加入20~50μL RNase-free Water或其他缓冲液溶解RNA沉淀。
- -80℃保存。
- 加入160μL RNase-free Water将产物稀释至180μL。
- 柱纯化
柱纯化可以去除蛋白和游离核苷酸。
纯化前加入80μL RNase-free Water将产物稀释至100μL,再按柱纯化说明书进行纯化。
注:由于RNA产量较高,为避免超出结合柱的承载能力,请对所需柱子数量进行预估。 - 磁珠纯化
磁珠纯化可以去除蛋白和游离核苷酸。
按磁珠纯化说明书进行纯化。 - 氯化锂纯化
- 20μL体积分别加入30μL的氯化锂沉淀液(7.5M氯化锂,50mM EDTA)和30μL RNase-Free Water(氯化锂的终浓度需保持在2.5-2.8M);
- 混匀后放入-20℃放置至少30min。
- 12000rpm离心15min,去上清,收集沉淀。
- 用预冷的70%的乙醇洗三次。
- RNase-Free Water复溶后检测。
- 20μL体积分别加入30μL的氯化锂沉淀液(7.5M氯化锂,50mM EDTA)和30μL RNase-Free Water(氯化锂的终浓度需保持在2.5-2.8M);
- 酚/氯仿纯化法
- RNA定量
- 紫外吸收法:游离核苷酸会影响定量的准确性,采用此方法前请先进行RNA纯化。
- 染料法:用RiboGreen染料进行RNA定量,游离核苷酸不会影响定量,可以对纯化或者未纯化的反应产物中的RNA进行准确定量。
- 紫外吸收法:游离核苷酸会影响定量的准确性,采用此方法前请先进行RNA纯化。

- Poly(A)尾比预期的长。
- 适当缩短反应时长;
- 适当减少反应体系中酶的加量;
- 通过引入更多的RNA来提高反应体系中RNA的浓度,或减小整个反应体系的体积。
- 适当缩短反应时长;
- Poly(A)尾比预期的短。
- 适当延长反应时间;
- 适当增加反应体系中酶的加量;
- 通过减少RNA的量来降低反应体系中RNA的浓度,或增大整个反应体系的体积。
- 适当延长反应时间;
- 加尾失败
- RNA 3’末端可能隐藏在二级结构中,可通过适当加热以解除二级结构影响;
- RNA底物被污染,可再次进行RNA底物纯化;
- 酶失活,确认酶是否保存于-20℃,实验操作时保持酶始终置于冰上;
- ATP降解,确认ATP保存于-20℃,验操作时保持酶始终置于冰上。
- RNA 3’末端可能隐藏在二级结构中,可通过适当加热以解除二级结构影响;
相关搜索:Poly(A)聚合酶,RNA加尾酶,RNA加A尾酶,RNA加尾试剂盒,E. coli Poly(A)Polymerase



